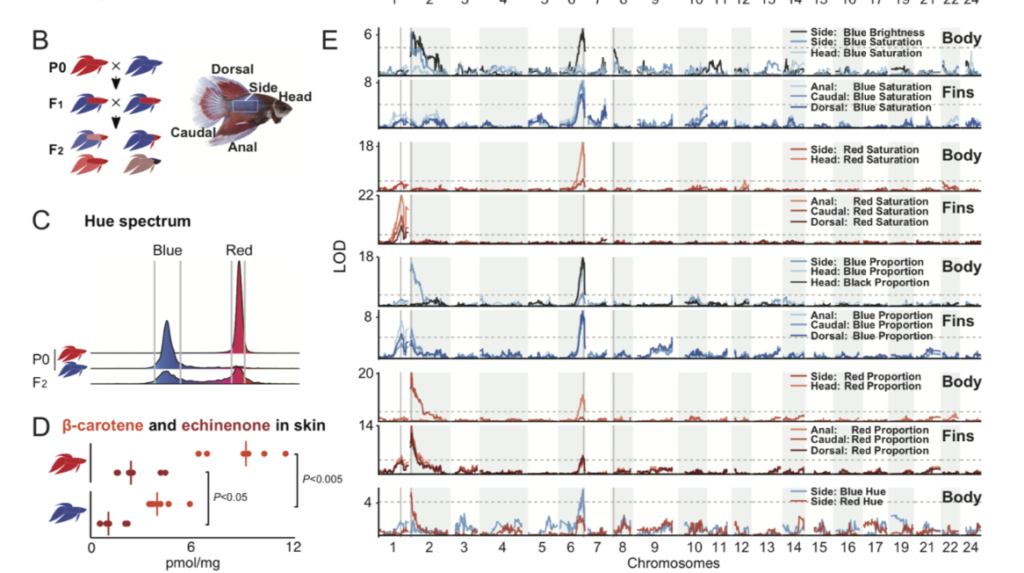
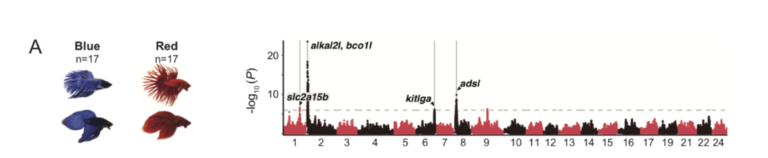
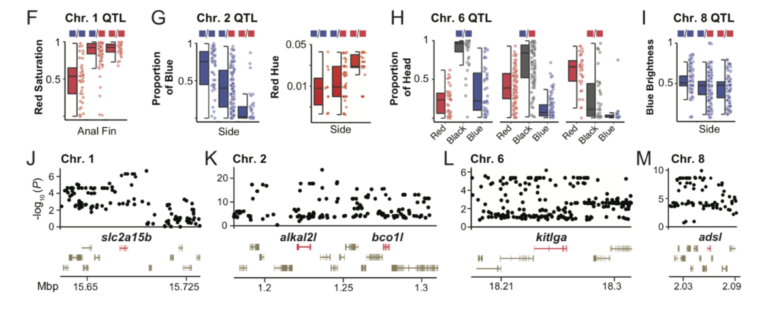
Un recente studio, pubblicato il 30 Aprile 2021, realizzato da Young Mi Kwon e colleghi, ha analizzato quali fossero le conseguenze dell’allevamento di selezione di Betta. Parte dello studio era volto a stabilire se la selezione fenotipica di alcune colorazione fosse correlata a dei geni in particolare. Negli anni gli allevatori di Betta sono riusciti a selezionare tantissime varietà diverse, dai moltissimi colori. Per poter studiare la correlazione tra geni e colori, gli studiosi hanno eseguito uno studio GWAS (genome–wide association study). Lo studio è un’analisi completa di tutti i geni di gruppi di individui, per poter vedere le differenze genetiche. Hanno utilizzato due gruppi di 17 esemplari ciascuno, un gruppo di esemplari rossi ed un gruppo di esemplari blu. Sono stati scelti esemplari rossi e blu perché si trovano agli opposti dello spettro di tonalità e perché differiscono anche per luminosità (il blu lo è, il rosso no). Dato che rosso e blu sono colori fissati, per poter avere un quadro più completo hanno incrociato esemplari rossi con esemplari blu ed hanno analizzato il genoma dei figli. Gli studiosi hanno misurato nei piccoli della F2 (211 esemplari) la proporzione di colore rosso, blu e scuro nelle pinne dorsali, anali e caudali e delle zone laterali del corpo e la testa. Lo studio GWAS ha indicato dei picchi in due loci nel cromosoma 2 : il augmentator-α2 (alkal2l) ed il beta-carotene oxygenase 1-like (bco1l). Queste regioni hanno mostrato una grande differenza tra il gruppo di Betta rossi ed il gruppo di Betta blu. Il alkal2l modula la saturazione del colore blu sul corpo, ma non su pinne e testa. La regione alkal2l codifica una ligando della tirosin-chinasi leucocitaria che viene espressa nei precursori degli iridofori, i cromatofori (cellule) che generano colori rifrangenti come il blu. Il bco1l codifica per una proteina che metabolizza il colore carotenoide rosso-arancio. Variazioni nel locus contenente alkal2l e bco1l probabilmente influenzano sia la colorazione blu che quella rossa attraverso questi due geni localizzati solo ~ 50 kb di distanza. Lo stretto collegamento potrebbe spiegare perché gli allevatori faticano a realizzare il pesci rossi “perfetti” senza alcuna iridescenza. Il secondo picco GWAS più elevato è stato trovato sul cromosoma 8, il adenylosuccinate lyase (adsl). Questo locus codifica un enzima coinvolto nella sintesi de novo delle purine, ovvero nella lucentezza del colore blu nel corpo. Le purine sono i componenti principali delle piastrine riflettenti negli iridofori della pelle dei pesci, e sono alla base dell’iridescenza. Queste piastrine differiscono nella loro struttura tra i Betta blu ed i Betta rossi. Il terzo picco GWAS più elevato, è stato trovato sul cromosoma 1, solute carrier family 2, member 15b (slc2a15b), un gene necessario per lo sviluppo di xantofori (cromatofori per il colore giallo) già descritto per i piccoli di medaka, ma il cui ruolo nella pigmentazione degli adulti non era stato precedentemente descritto. Il slc2a15b influenza fortemente la saturazione del rosso nelle pinne, ma nel corpo o nella testa. La colorazione intensa delle pinne rispetto al corpo è un fenotipo chiamato dagli allevatori come varietà “cambodian”, e i risultati di questo studio suggeriscono che slc2a15b contribuisca a questo fenotipo. Il quarto picco GWAS più elevato, trovato sul cromosoma 6, il kit ligand (kitlga). Gli ortologhi di questo locus sono stati già studiati ed è stato dimostrato che influenzano la pigmentazione della melanina in altri pesci e nei mammiferi. Il kitlga influenza fortemente la proporzione di nero, blu e rosso sulla testa e sulle pinne, ma meno sul corpo.La testa nera è un fenotipo che abbiamo trovato essere legato a kitlga. Gli allevatori di Betta chiamano questa colorazione, la testa nera, “mask”. L’impatto relativamente più forte sulla saturazione del blu potrebbe essere relativo alla stretta associazione istologica di iridofori e melanofori nella struttura epidermica del Betta.

Fonte dell’articolo:
•Genomic consequences of domestication of the Siamese fighting fish, Young Mi Kwon et al., 2021. https://doi.org/10.1101/2021.04.29.442030